【テキストマイニング】 bigram を用いて小説の書き手の識別をしてみる
公開日:
:
最終更新日:2018/11/26
テキストマイニング bigram, R, RMeCab, クラスター分析, テキストマイニング入門, デンドログラム, 小説, 形態素解析, 書き手の識別
「テキストマイニング入門」という本に書かれている内容に添って RMeCab を使った小説の分析を行ってみます。
テキストマイニング入門とは、徳島大学の石田基広氏が書いた主にRとそのライブラリを用いたテキストマイニングの入門書です。
フリーの統計ソフトのRのコマンドが実際に書かれており、さらに本書で使用されたデータもダウンロードできます。
丁寧に解説していて初心者でも非常に分かりやすくおすすめです。
※ R と RMeCab をインストールしてから分析を始めます。
Contents
分析対象データ
今回は、森鴎外と夏目漱石の小説各4編づつの合計8編を分析対象としています。
使用データセットは以下のURLからダウンロードできます。
Windows用 Shif-Jis コード:
http://web.ias.tokushima-u.ac.jp/linguistik/RMeCab/data.zipMac / Linux 用 UTF8 コード:
http://web.ias.tokushima-u.ac.jp/linguistik/RMeCab/data.tar.gz
今回用いるデータは data/writers ディレクトリの直下にあります。
WindowsとMac/Linuxは文字コードが違うため入れ替えてしまうと文字化けしてしまいます。
また、このデータには文字数をそろえるように、それぞれ1万6000語で切っているようです。
bigram【バイグラム】でクラスター分析
事前に、以下のコマンドでRMeCabライブラリを読み込んでおきます。
|
1 |
library(RMeCab) |
まず、R上でカレントディレクトリを確認します。
|
1 |
getwd() |
カレントディレクトリの直下に解凍した data ディレクトリをコピーします。
次に、docNgram関数を使用します。
docNgramはファイルを一括してNgramを解析できる関数です。
引数にディレクトリを指定すると、指定したディレクトリ以下に含まれる全てのファイルが対象となります。
type=0 で、文字単位のバイグラムを返します。
|
1 |
res <- docNgram("data/writers", type=0) |
type=1 で、形態素単位のバイグラムを返します。
|
1 |
res <- docNgram("data/writers", type=1) |
type=2 で、品詞情単位のバイグラムを返します。
|
1 |
res <- docNgram("data/writers", type=2) |
今回は、type=0 の文字単位のバイグラムを使います。
最終的にクラスター分析で似ている(距離が近い)小説同士をくっつけていきたいのですが、
どれくらい似ているか、というのは具体的な数値(距離)が必要です。
一般的な距離の尺度としては、ユークリッド距離が用いられます。
ここで、バイグラムのデータに戻りますが、現状は
- 行にバイグラムの出現頻度(出現がなければ0が入る)
- 列に8つの小説
をとる行列になっています。
行と列の総数の確認は以下のコマンドで行います。
|
1 2 |
> nrow(res) #行数を確認 [1] 26541 |
|
1 2 |
> ncol(res) #列数を確認 [1] 8 |
次にユークリッド距離を求める dist() 関数を使います。
ユークリッド距離では、2点間の距離を求めて、それらを全て加算したものと言えますが、
2点間の距離は、
それぞれのバイグラムの頻度(例えばどちらも出現しない=0の場合はユークリッド距離に影響ないが、片方にのみ1回出現している場合は距離は1となりユークリッド距離に影響が出てきます)から求めます。
ここで注意しないといけないことがあり、ユークリッド距離 dist を使うためには res の行と列を以下のようにして入れ替えなければなりません。
|
1 |
t(res) |
この処理を転値といいます。転値後、distに値を渡すと、
|
1 |
dist(t(res)) |
以下のような結果になり、それぞれの小説間での距離が求められていることがわかります。
|
1 2 3 4 5 6 7 8 |
ogai_gan.txt ogai_kanoyoni.txt ogai_niwatori.txt ogai_vita.txt soseki_eijitsu.txt soseki_garasu.txt soseki_omoidasu.txt ogai_kanoyoni.txt 319.3509 ogai_niwatori.txt 392.3340 421.0285 ogai_vita.txt 376.8302 441.0397 373.3243 soseki_eijitsu.txt 366.6742 418.6944 413.7971 402.2624 soseki_garasu.txt 447.9520 496.6528 512.4246 452.5086 396.0568 soseki_omoidasu.txt 355.6473 423.2989 460.9176 403.1030 349.1031 375.3159 soseki_yume.txt 410.5801 462.7170 454.5283 442.7561 267.1030 380.3380 392.0191 |
距離が求まったので、この値を使ってクラスター分析を行います。
今回は、ウォード法を用います。
|
1 |
hclust(dist(t(res)),"ward") |
このままでは、結果がよくわからないので、plot()関数を用いて可視化します。
|
1 |
plot(hclust(dist(t(res)),"ward")) |
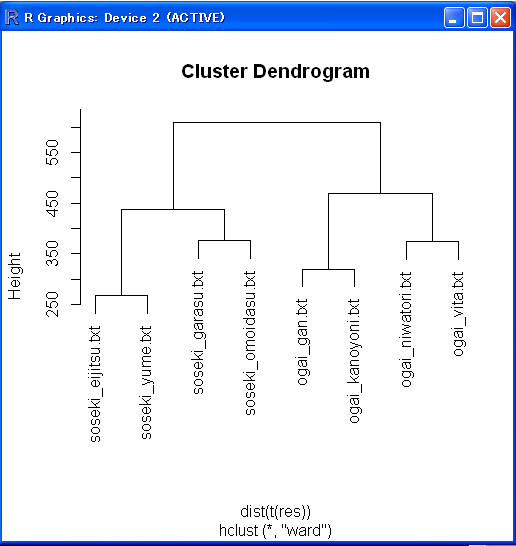
すると、このようにデンドログラムが描画されます。
森鴎外と夏目漱石がきれいに分離されていることが分かります。
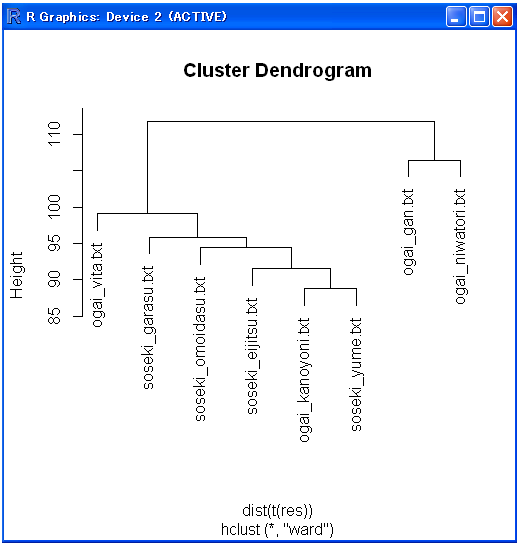
バイグラム(単語・形態素単位)でも検証
本書では、文字単位でのバイグラムを用いていたが、
単語・形態素単位のバイグラムではどうなるのか検証してみました。
docNgram()関数で type=1 とすると単語・形態素単位のバイグラムになります。
|
1 |
res <- docNgram("data/writers", type=1) |
res にはこのようなバイグラムの頻度が入ります。
行数を確認してみます。
|
1 2 |
> nrow(res) #行数を確認 [1] 21806 |
文字単位のバイグラムより約5千要素ほど少なくなっています。
そして同様に、ユークリッド距離を求めて、クラスター分析をしてみました。
関連記事
-

-
【Python】NLTK(自然言語処理ライブラリ)を使ってみた。
Python の自然言語処理ライブラリである NLTK を Linux 環境にインストールして使って
-

-
【Python】形態素解析器 Mecab を Linux(Ubuntu)で使ってみた。
Linux の Ubuntu OS に、形態素解析器 Mecab をインストールし、使ってみたときの
-

-
【mecab-python3】parseToNode で surface が正しく取得できないときの対処法。
python3 で mecab-python3 を使うと、parseToNode で surface
- PREV
- 【Gmailアドレス】を複数取得する方法
- NEXT
- 税理士試験の受験資格と税理士になる方法を整理する